서 론
치주질환은 치주조직의 염증과 구조의 소실을 야기시키는 질환으로 다양한 전신적 및 국소적 요인들에 의하여 발병되며, 주원인은 치면세균막 내 혐기성 세균이다. Sastri [1]는 치면세균막 내에 약 400여 세균 종들이 존재한다고 보고하였고, 현재는 특정 치주질환과 관련된 구강 내 병원성 세균간의 역학관계 및 치주질환 병소에 호발하는 세균의 분포양상에 관한 연구들이 활발히 이루어지고 있다.
치주질환 중 가장 흔한 성인형 치주염은 치은연하 치면세균막 내의 Porphyromonas gingivalis (P. gingivalis) 등 7종의 세균과 깊은 관련성이 있다[2]. 질환의 정도에 따라 병원성 세균의 종류나 그 총량의 차이를 보이며, 건강한 사람 간에서도 전신건강이나 면역력에서 유의한 차이가 나타나기도 한다. 균 서식지의 특성으로서 대부분의 치주질환균은 산소에 민감한 혐기성 세균들이기 때문에 치은연상 치면세균막에 비해 산소의 공급이 원활하지 못한 치은연하 치면세균막에 분포한다[3]. 그러나 최근에는 치은연상 치면세균막은 치은연하 치면세균막과 서로 연결되어 있으며, 장기간의 침착으로 두꺼워지면서 공기의 유입이 차단됨으로써 혐기성 세균이 존재할 가능성이 보고되면서 치은연상 치면세균막에 있는 치주질환 관련 세균에 대한 관심이 높아지고 있다.
Actinobacillus actinomycetemcomitans (A. actinomycetemcomitans), P. gingivalis, Prevotella intermedius (P. intermedius)는 3대 치주질환균으로 널리 알려져 있고, 치은염에서 파괴적인 치주염으로의 전이기에 관여하는 F. nucleatum은 치은열구에서 주로 발견되며 다른 주요 세균과의 공동 응집에 중심적 역할을 한다[4]. 단위부피당 형성된 집락 수(colony forming unit, CFU)를 측정하는 방법으로 치주질환 병소부위에 존재하는 원인균을 탐색하였으나 치주질환 여부와 관계없이 원인균이 발견되지 않는 경우도 있었다. 더욱이 CFU법은 특정 목표 균을 찾거나 배양하기 위해 선택적 배지를 만들어야 하는데, 아직까지 구강 내 일부 균들에 대한 선택배지만이 정량 및 정성적으로 확인하도록 확립되었을 뿐이다[5,6].
따라서 1990년대 중반부터는 분자생물학의 발전으로 25-100개의 세균만 있어도 검출이 가능한 중합효소연쇄반응(polymerase chain reaction, PCR)이 사용되고 있다[7]. PCR은 세균 특이성이 있는 primer를 이용하여 적은 수의 세균이 있을지라도 쉽게 검출할 수 있는 유용한 방법이며, 이를 이용하여 구강 내 치면세균막이나 타액에서 직접 세균을 검출할 수 있게 되었다[8]. 타액을 대상으로 PCR분석을 할 경우 타액 내에 들어있는 화합물들이 PCR 증폭에 영향을 주어 우려도 있지만[9,10], 그러한 요인을 제거하는 Chelex 100을 이용한 깨끗한 DNA를 추출하는 방법 등으로 구강 내 타액의 복잡한 환경에 의해 분석이 용이하지 못한 부분을 해소하고 있다[11]. 이러한 방법을 이용하여 치주질환 원인 균에 대한 정성적인 연구에 많이 활용되고 있지만 PCR은 중합반응의 최종 단계를 알아내는 것으로써 목표 균의 존재여부를 조사할 수 있는 반면 정량적 계산은 여전히 어렵다.
그러나 최근에 실시간 중합효소연쇄반응(real-time polymerase chain reaction, RT-PCR)이 확립되어 지수적 증식 단계를 조사함으로써 원인 균의 존재여부를 파악할뿐 아니라 정량적 분석까지도 가능하게 되었다[12]. 선행연구에 따르면, 전통적 중합효소연쇄반응(c-PCR)과 RT-PCR을 이용한 연구에서 타액과 치은연하 치면세균막에서 치주세균의 출현율은 높은 일치성을 보였으며, 특히 RT-PCR은 총 분석시간이 40분 정도로 시간을 단축할 수 있는 것으로 밝혀졌다[13]. 따라서 최근에는 보편적으로 임상적인 미생물의 총량 분석방법이 RT-PCR법으로 확립되었으며, 미국의 국립의학중앙도서관(National Library of Medicine National Institutes of Health, NCBI)에서 미생물로부터 DNA추출법과 Probe method 등이 표준화되어 등록됨으로써 신뢰성을 확보하였다[14,15]. 아울러 RT-PCR을 응용하여 한 번에 다종의 균을 정량분석 할 수 있도록 실시간 다중중합효소연쇄반응(multiplex real-time polymerase chain reaction, MRT-PCR)법이 소개되고 있다.
본 연구의 목적은 선행 연구를 기반으로 다종의 치주질환 원인 균을 짧은 시간에 분석 가능한 MRT-PCR법의 구강미생물 정량분석에 대한 정확성을 확인하기 위함이다. 이를 위해 선택배지가 정형화된 치주질환 원인균인 P. intermedia와 치아우식 원인균인 Streptococcus mutans (S. mutans)에 대한 전체 세균의 수를 확인하는 CFU법과 비교분석하여 MRT-PCR법의 유용성을 평가하였다.
연구방법
1. 연구대상 및 시료채취
2016년 9월 20일에서 10월 18일까지 치주질환 관련 진료를 위해 C대학교 치과병원 치주과에 내원한 20세에서 65세 사이의 환자 30명을 대상자로 정하였으며 평균 연령은 44세이었다. 표본 수는 G*power 3.1.9.2를 이용하여 효과크기=0.5, 유의수준 α=0.05, power=0.95로 정하여 산출한 결과 28명이었다.
선정된 연구대상자들에게 본 실험의 개요를 설명하고, 연구의 취지에 적극적으로 동의한 자를 최종적으로 대상자로 정하였다(IRB No. CUDHIRB-1609-004). 대상자의 선정기준은 치은염관리가 필요한 치은염지수[16]가 최소 1이상인 가운데, 임신하거나 수유 중이거나 치주질환 진행에 영향을 줄만한 전신적인 상태에 있거나, 3개월 내에 항생제 치료를 받은 경우에는 제외하였다.
치과전문의 1인과 치과위생사로 구성된 검체 수집 팀은 모든 연구대상자를 대상으로 치아 위치 별로 치주질환 정도를 조사했으며, 전신질환 등을 포함하여 임상증례서에 기록하였다. 연구대상자에게는 개인 위생관리 내용 등의 편차나 하루 일과 중의 오차 등을 최소화하기 위해 오전 11시에 일괄적으로 20 ml의 멸균된 증류수로 30초간 강하게 가글하고, 이를 50 ml conical tube에 뱉도록 하여 타액을 채취하여 급속 냉동보관하여 시료로 사용하였다. 이때 균들의 환경적 변화요소로 인한 균의 변화나 변수를 최소화하기 위해 검체 수집 후 준비된 선택배지에 균 심기와 MRT-PCR로 즉시 분석하였다.
2. 세균 및 세균배양
본 연구에서 사용된 가글 한 타액 검체는 100 ul이며 전체세균 및 치주질환과 치아우식에 관여하는 P. intermedia와 S. mutans의 배양을 위해 사용된 배지조건 전체 세균 대상으로는 1% tryptic soy broth, 0.5% yeast extract, 0.5% NaCl, 0.2% glucose, 0.02% L-tryptophan, 1.5% Bacto agar, 5% defibrinized sheep blood, 4 ul/ml erythromycin, 5 ul/ml crystal violet[17], P. intermedia는 3% Tryptic soy broth 0.5% yeast extract, 0.05% cysteine HCl-H2O, 0.5 mg/mlhemin 및 2 ug/ml vitamin K1이 배합되었다[18].
S. mutans는 Mitis Salivarys Agar (DiffcoTM, USA)에 0.1% K-tellurite (SIGMA, USA)와 0.2 unit/ml bactracin (SIGMA, USA)이 배합된 배지에 도말하였다[19]. 그 다음 37°C 혐기성 배양기(85% N2, 5% CO2, 10% H2, Model bactron I, Cornelius, OR, USA)에서 배양하였다. 이때 20 ml의 멸균 증류수로 가글한 타액을 1,000배, 10,000배, 100,000배 희석하였다. 상기 기술한 세균 배지에 도말 한 다음, 배지는 혐기성 배양기에서 7일 동안 배양한 후 실험에 사용하였다.
3. 구강내 미생물 총량 분석
1) CFU법
7일 동안 배양 후 즉시 집락 계산기를 사용하여 생성된 집락 수를 산출하였다. 부득이할 경우에는 4°C에 보존시켜 24시간 이내에 산정하였으며 집락 수의 계산은 확산 집락이 없고(전면의 1/2 이하 일 때에는 지장이 없음) 1개의 배지에 30~300개의 집락을 생성한 배지를 택하여 집락 수를 계산하는 것을 원칙으로 하였다. 배지에 300개 이상 집락이 발생한 경우 300에 가까운 평판에 대하여 CFU법에 따라 반지름이 9 cm의 petri dish인 경우에는 1 cm2내의 평판 집락 수에 65를 곱하여 그 평판의 집락 수로 계산하고 전 평판의 30개 이하의 집락만을 얻었을 경우에는 가장 희석배수가 낮은 것을 측정하였다.
2) Genomic DNA추출
표준균주 및 검체 시료는 1 ml를 기준으로 NEXampTM Easy Direct-PCR Buffer (Geneslab co. Gyeonggi-do, Korea)를 이용하여 genomic DNA를 추출하였다. 다음으로 시료를 12,000 rpm 10분 동안 원심 분리하여 상등액은 제거하고 100 ul direct-PCR Buffer를 넣고 잘 혼합한 다음 상온에 10 분간 방치하였다. 그 이후 12,000 rpm에서 3분간 원심분리 한 후 상등액을 새로운 Eppendorf tube에 넣어 PCR에 사용하였고, 즉시 사용하지 않을 경우 -50°C 이하 냉동 보관하였다.
3) Multiplex Real-time PCR법
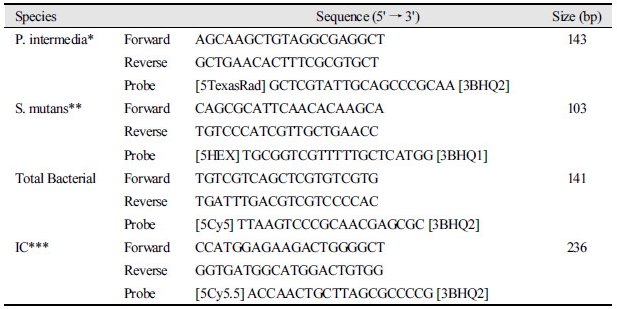
MRT-PCR에 사용된 probe는 Taqman probe방식으로 이용되며[20], probe 형광 간의 간섭을 고려하여 서로 파장이 겹치지 않게 2종 선정하였다. 각 반응에 Total bacteria 및 Internal control을 동시에 분석하게 구성하였으며 Total bacterial primer & probe는 NCBI data base에 등록된 16s rRNA gene을 ‘Multiple Sequence Alignment by CLUSTALW web program’으로 분석하여 P. intermedia와 S. mutans 및 전체 세균의 총량을 분석하였다[15,21]. 이와 함께 검체 DNA의 포함 여부 및 MRT-PCR 반응 적정성을 확인하기 위해 GAPDH gene을 바탕으로 primer & probe를 위와 동일한 방법을 이용하여 internal control로 사용하였다[15,21]<Table 1>.
|
Table 1. Primer and probe used for multiplex real-time PCR
|
|
|
*Prevotella intermedia, **Streptococcus mutans, ***Internal control |
|
MRT-PCR과 결과 분석은 CFX 96 touchTM real-time PCR detection system (Bio-Rad Co., California, U.S.A)를 이용하였다. Cycle threshold (Ct) value는 ‘CFX ManagerTM Software v3.1’에 의해 산출하였다. 3 ul의 추출된 genomic DNA에 12.5 ul의 HS prime qPCR premix (Jenetbio Co., Daejeon, Korea), Forward & reverse primer는 각 1 uM, probe는 0.5 uM이 되도록 첨가하였으며 멸균 증류수로 최종 25 ul의 PCR 혼합용액을 만들었다.
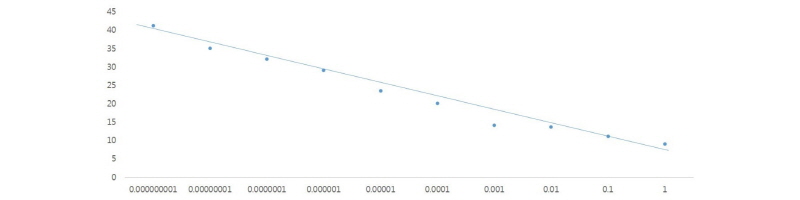
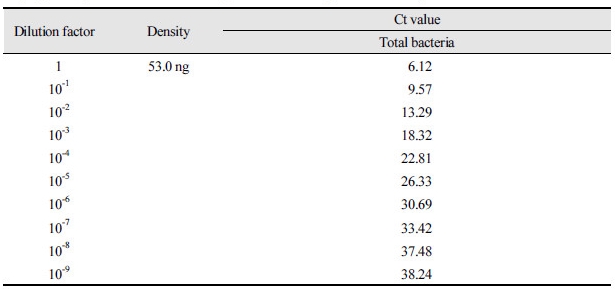
PCR의 반응조건은 95°C에서 10분, 95°C에서 10초, 63°C에서 30초로 45회 반복하여 수행하였다. 이때 실시간 핵산 증폭 결과인 Ct값에 따른 핵산의 농도를 결정하기 위하여 Nucleic acid spectrophotometer (ACT gene Inc., USA)를 사용하여 260 nm에서 측정한 흡광도를 plasmid DNA copy 수로 환산하였다. 109 Copy의 plasmid를 10 fold serial dilution하여 Ct값에 따른 plasmid DNA copy number의 표준곡선을 확인한 후 시료들의 정량에 적용하였으며, Total bacteria 프라이머 및 프로브의 최소검출범위를 측정하기 위해 P. gingivalis (KCOM3001)의 유전체 DNA를 101부터 1010까지 10단계씩 연속적으로 희석하여 표준 곡선을 만들었다<Fig. 1><Table 2>.
|
|
|
|
Fig. 1. A standard curve was generated from the amplification plot of P. gingivalis. A suspension of P. gingivalis plasmid DNA was serially diluted and subjected to multiplex real-time PCR |
|
|
Table 2. A standard curve was generated from the amplification plot of P. gingivalis. A suspension of P. gingivalis plasmid DNA was serially diluted and subjected to multiplex real-time PCR
|
4) 통계분석
IBM SPSS 23.0 (IBM Corp., Armonk, NY, USA)을 이용하여 수집된 자료의 빈도와 평균 및 표준편차를 구하였다. 성별과 연령별 MRT-PCR에 의한 치은염지수의 차이를 Independent t-test와 One way ANOVA 분석을 시행하였고, CPU법과 MRT-PCR법에 의한 전체 세균 량 비교는 Independent t-test로 분석하였다. 유의수준은 α=0.05로 정하였다.
연구결과
1. 대상자의 인구학적 특성에 대한 동질성 검정
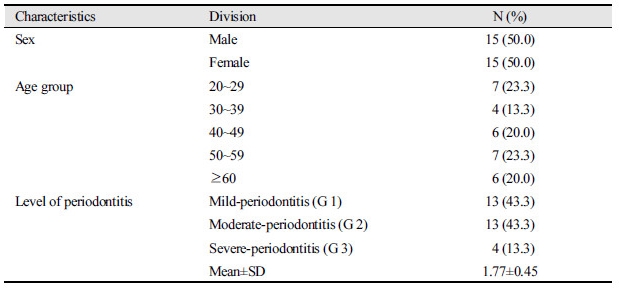
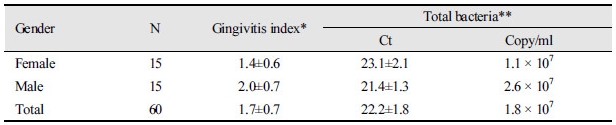
MRT-PCR법을 활용하여 C대학교 치과병원 치주과에 내원한 총 30명의 피검자에 대한 Silness- Löe의 치은염지수를 조사한 결과<Table 3>, 치은염지수가 평균 1.77의 중등도인 치은염을 가졌다.
<Table 4>는 성별에 따른 치은염 지수와 전체 세균양을 분석한 결과이다. 성별로는 여성 (1.4±0.6)이 남성(2.0±0.7)에 비해 치은염이 유의하게 높았고(p<0.05), 이에 따른 전체 세균양은 남성이 높은 수준이었으나 통계적으로 유의한 차이는 없었다(p>0.05).
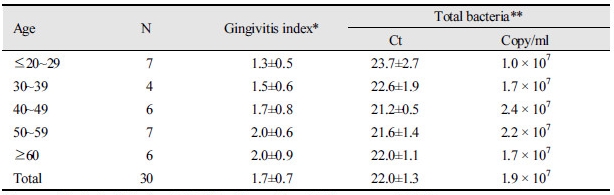
<Table 5>는 연령별에 따른 치은염 정도를 나타내며, 연령이 증가할수록 치은염이 높았으나 구강 내 전체 세균 양에 대한 통계학적 차이는 나타나지 않았다(p>0.05).
|
Table 5. Prevalence of total bacteria in age group by MRT-PCR Units: Mean±SD
|
|
|
*Age-specific gingival index is p<0.05, **Total bacteria is p>0.05 by one-way ANOVA test |
|
2. 치은염 지수 별 구강세균의 총량
<Table 6>은 MRT-PCR법을 이용하여 치은염 단계별 구강 내 전체세균 양을 조사한 결과이다. 초기 치은염으로 조사된 대상자의 경우 구강 내 전체세균 양은 1.1 × 106 Copy/ml로 확인되었으며, 질환의 정도가 심할수록 증가하여 통계적으로 유의하게 나타났다(p<0.05).
3. MRT-PCR법과 CFU법 비교
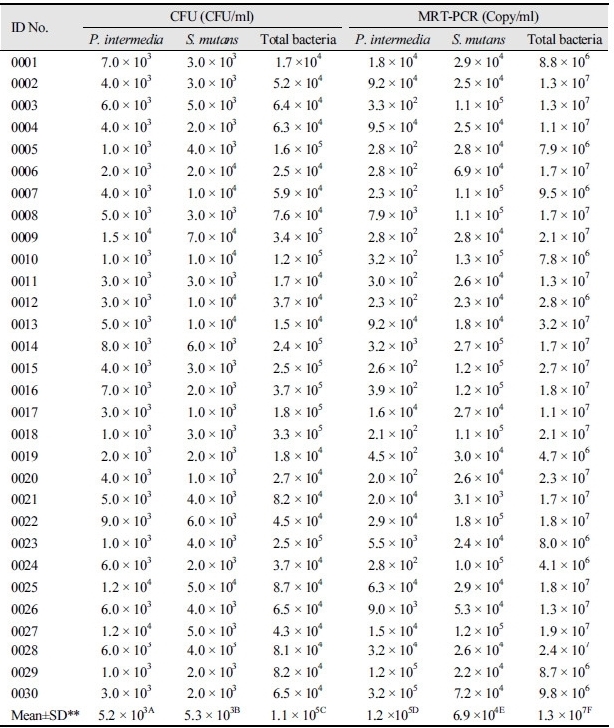
전체 세균 량에 대하여 CFU법은 최소 ~ × 106 CFU/ml에서 최대 ~ × 107 CFU/ml 미생물 수가 확인된 반면에 MRT-PCR법에서는 최소 ~ × 106 Copy/ml에서 최대 ~ × 107 Copy/ml 미생물 수를 확인하였다. 평균적으로 CFU법은 1.0 × 105 CFU/ml이었고, MRT-PCR법은 1.3 × 107 Copy/ml이었다. 이는 두 정량 법 비교를 통해 살아있는 미생물만 배양되어 수를 확인하는 CFU법의 결과와 MRT- PCR법에 의한 미생물의 수에서 유의한 차이가 있는 것으로 밝혀졌다.
치주질환 원인 미생물 P. intermedia 및 치아우식 원인 미생물 S. mutans 에 대해서도 동일하게 진행한 결과, CFU법에서 P. intermedia 및 S. mutans는 각각 평균 ~ × 103 CFU/ml와 ~ × 103 CFU/ml로 나타났으며 MRT-PCR법에서는 ~ × 105 CFU/ml와 ~ × 104 CFU/ml로 전체 세균 량 결과와 유사한 결과로 유의한 차이가 있었다<Table 7>.
총괄 및 고안
과거에는 문헌과 연구에서는 구강미생물의 종류는 400여개 정도로 가름하였으나 PCR을 통한 유전자 염기서열 분석법으로 추가 300여종 이상이 발견되고 있으며, 현재도 그 종류는 지속적으로 발견되어 700여종에 이른다. 이러한 미생물의 속에서도 다양한 종이 발견되는 과학적 진보의 결과다. 한편 전통적으로 선택적 배지를 활용하여 특정 세균만을 배양하는 방법으로서 미생물 분석 표준 정량법으로 알려졌다[22]. 그러나 CFU법은 잘못된 결과를 얻을 수 있고[17,23], 현재까지 모든 미생물의 선택적 배지가 개발되지 않았고, 구강미생물 700여종 가운데, 불과 20여개의 선택적 배지법이 규명되었다. 따라서 이미 균 배양이 정립된 선택배지를 통하여 배양 가능한 구강미생물 중 대표적인 치아우식 균인 S. mutans와 치주질환 균 P. intermedia를 정량화하여 MRT-PCR법과 비교분석하였다. 한편, PCR법에 의한 정량분석의 특징은 구강 내 사멸된 균에서도 DNA 추출 정량인 만큼 일반적으로 살아있는 미생물을 대상으로 한 선택배지 군락형성으로 인한 균 정량법보다 많은 균량이 검출되는 것이 일반적이다[24].
이러한 PCR법에 의한 미생물 총량의 정보는 CFU법에 의한 군집 수를 배제한다면, 구강 내 사멸 균의 총량을 확인할 수 있는 방법으로도 사용 가능하다. 구강 내 사멸 균 정량도 중요한 까닭은 치주질환 유발 균은 특성상 혐기성세균으로서 자신의 세포외막에 강력한 독성물질인 지질다당체(lipopolysaccharide, LPS)로 둘러 쌓여 있으며, fimbriata 등 이들 세포의 부속물질들도 치주조직에 박히거나 염증물질을 방출하여 치주질환을 발생시키는데 살아있는 균만큼의 독성을 유지하기 때문이다[25]. 따라서 사멸균일지라도 구강질환에서 특히 치주질환을 예방하기 위해서는 적당한 구강관리를 통하여 구강 내 살아있는 균의 억제뿐 아니라 사멸 균도 최소화해야 한다.
본 연구에서는 이러한 MRT-PCR의 특징을 활용하여, CFU법과 MRT-PCR법으로 정량 비교분석한 결과 전체 세균 량에 대하여 CFU법은 최소 ~ × 106 CFU/ml에서 최대 ~ × 107 CFU/ml의 미생물 수가 확인된 점에 비해 MRT-PCR법으로 분석한 결과에서는 최소 ~ × 106 Copy/ml에서 최대 ~ × 107 Copy/ml로 높게 나타났다. 즉, 평균적으로 CFU법은 1.0 × 105 CFU/ml이었고, MRT-PCR법은 1.3 × 107 Copy/ml로 나타난 결과는 살아있는 미생물만 배양되어 수를 확인하는 CFU법 보다는 MRT-PCR법으로는 더 세밀하게 구강 내 세균이 나타나는 것을 확인할 수 있었다. 치주질환을 유발균인 P. intermedia 및 치아우식 유발균인 S. mutans에 대해서도 동일하게 진행한 결과, CFU법에서 P. intermedia 및 S. mutans는 각각 평균 ~ × 103 CFU/ml과 ~ × 103 CFU/ml로 나타났고, MRT-PCR법에서는 ~ × 105 CFU/ml와 ~ × 104 CFU/ml로 전체 세균 량 결과와 마찬가지로 통계적으로 유의한 차이가 있었다. 이와 유사한 연구결과로 치면세균막 내 특정 세균 검출을 비교한 연구를 통해 CFU법과 PCR법은 유의한 차이가 있었으며[26], 근관 및 치주 미생물학에 대한 CFU법과 PCR법을 비교하여 특정 세균의 검출은 CFU법보다 PCR법이 더 민감하고 높은 신뢰성이 있다는 결과를 확인할 수 있었다[27-30]. 따라서 두 정량 법 비교를 통해 살아있는 미생물만 배양되어 수를 확인하는 CFU법의 결과를 기준으로 MRT-PCR 결과 미생물 수의 차이가 유의하였으며 기존 연구에서도 qPCR과 Fish 혹은 Immunofluorescence CFU법으로 비교 시 훨씬 낮은 수치로 세균 수가 나타났다[31]. 또 다른 RT-PCR법과 CFU법의 비교분석 연구에서도 CFU법에서는 산소에 민감한 T. denticola를 검출하지 못했지만 RT-PCR법에서는 검출이 되었으며, 이는 형광첨자(probe)를 이용하여 세균검출 수준이 매우 높은 것을 알 수 있다[23]. 또한 CFU법을 통해 세균을 배양할 경우 잘못된 결과를 산출 할 수도 있고, 특히 혐기성 배양은 시간이 오래 걸리고 힘들며 상대적으로 민감도가 PCR에 비해 낮았다[33].
이와 유사한 다른 연구에서 치아우식 균인 S. mutans를 RT-PCR법과 CFU법을 이용하여 정량 분석한 결과 RT-PCR법이 더 높은 정량이 나타나는 것을 확인하였고[34], 치주질환 균 P. intermedia는 RT-PCR법과 CFU법으로 비교한 결과 정량 값이 일치하지 않았으며[35], 이번 연구결과에서도 유사한 결과로 나타났다. 하지만 세균 수의 차이가 나는 이유는 CFU법은 산소에 민감한 혐기성세균일 경우 자라지 못하거나 죽은 세포까지 추적이 되었기 때문인데, 본 연구에서도 동일한 이유라고 판단된다. 앞서 언급했듯이 죽은 세포도 살아있는 세포와 같이 중요한 이유는 세포의 생사와 무관하게 세포외막의 독성, 세포날개 등이 치주조직에 염증을 지속적으로 야기시키는 병인이기 때문이다[36].
결론적으로 이번 연구의 목적은 대표적 치주질환 균과 치아우식 균을 선택배지로 배양한 CFU법에 의한 균의 총량과 새로운 미생물 총량을 동시 분석하는 MRT-PCR의 유용성을 조사하기 위한 것이었다. 선행연구들에 따르면, MRT-PCR방법은 사멸된 균까지 추가로 추적되어서 사멸 균의 위험성까지 경고할 수 있었다. 그러나 구강질환의 특징은 일반 전신의 감영성 질환과 달리 그 원인 균이 다양하다는 특징을 가졌다. 성 매개감염질환의 경우, 그 원인 균은 Human papilloma virus (HPV), Phthirus pubis (P. pubis), Herpes simplex virus (HSV), Chlamydia trachomatis (C. trachomatis), Treponema pallidum (T. pallidum), Trichomonas vaginalis (T. vaginalis), Neisseria gonorrhea (N. gonorrhea) 등 매우 다양한데, 이들은 각각의 임상적 특징이 달라서 각각의 성병으로 정의하고 있다. 아울러 이들 균은 특정인에게 발견된다면 성병에 감염되었다고 규정하고 임상적 증상이 대부분 발현되는 선별 기준치와 분류기준치라는 타당도 조사결과가 매우 높은 결과를 임상가나 연구가들이 얻을 수 있다. 다른 예로 폐렴의 원인 균은 pneumonia 속인데 이에 대한 아형 또한 다양하다. 그러나 이들 아형이 발견되면 폐렴 선별기준치로서 질병분류기준치에 대한 타당도도 매우 높게 나타난다는 특징을 가졌다. 그러나 구강 내 치아우식 균은 치아의 부위별 야기하는 곳이 다소 달라 S. mutans, Lactobacillus casei (L. casei), Actinomyces viscosus (A. viscosus), Actinomycetes naeslundii (A. naeslundii) 등을 원인 균으로 간주한다[37]. 하지만 이들 균들이 존재한다고 해서 반드시 치아우식을 야기하는 것은 아니다. 즉, 기회성 감염균으로 작용하는 경우가 많으며, 개인의 위생관리, 가족력, 면역 등 다양한 요소에 따라 치아우식 발생이 다르다. 치주질환 균도 마찬가지로 A. actinomy-cetemcomitans, P. gingivalis, Tannerella forsythensis (T. forsythensis), Treponema denticola (T. denticola), Capnocytophaga gingivalis (C. gingivalis), Fusobacterium nucleatum (F. nucleatum), Prevotella intermedia (P. intermedia), Eikenella corrodens (E. corrodens), Campylobacter rectus (C. rectus) 등이 치주질환 원인 균으로 인식되어지고 있다[18].
따라서 앞서 폐렴균이나 성 매개감염질환의 경우와 달리 치아우식이나 치주질환 균에 대한 연구에서 분류기준에 대한 선별기준치의 타당도를 비교할 수가 없다는 것이다. 그 원인질환의 종류도 다양하고, 종별다양성은 차치하고라도 그 속 별 균종이 다양하며, 또한 아직까지 정확히 각각의 균들이 질병에 대한 특징적 요소를 가지고 분류되지 않고 치아우식과 치주질환이라고만 정의하고 있기 때문이다. 이러한 이유로 본 연구에서는 MRT-PCR법의 타당도 조사에 따른 민감도나 특이도를 연구계획에 포함시키지는 못하였지만, 향후 구강미생물의 정량적, 정성적 분석에서는 고전적이고 배양할 수 있는 세균이 수종에 불과한 CFU법보다는 RT-PCR법이 널리 이용될 것으로 판단된다. 한편, 혐기성세균인 치은연하 세균의 조성에 가장 영향을 미치는 인자는 치주질환의 병기에 따라 달라지며, 만성 치주염을 가진 환자의 치은연하에서 T. forsythensis, P. gingivalis, T. denticola 같은 치주질환 원인 균이 평균적으로 양과 비율, 발생 빈도(prevalence)에서 증가한다고 보고되어 왔다[38]. 즉, 치주질환의 경우 분류기준치에 따른 임상적 병기에 따라 치주질환 균의 종류와 그 양이 변화한다는 것이다. 이러한 어려운 점 때문에 치아우식과 치주질환에 있어서 선별수치와 분류수치의 타당도 검사는 매우 어려운 현실이다[6]. 그러나 의료기관에서 빠르고 정확한 결과를 필요로 하기 때문에 MRT-PCR법은 건강한 사람을 대상으로 질병의 예상과 함께 환자로서 내원할 경우 치료 전과 후의 치료결과에 따른 모니터링 또는 스크리닝으로 매우 유효한 방법일 것이다. 향후 이러한 연구설계는 지속적으로 추적 연구가 시행되어야 할 것이다.
결 론
최근 기술 향상으로 한번에 여러 종의 세균을 분석할 수 있는 MRT-PCR이 개발되었기에 본 연구에서는 선택배지가 정형화된 치주질환 원인균인 P. intermedia과 치아우식 원인균인 S. mutans를 대상으로 국내에서 아직까지 통용되는 CFU법과 MRT-PCR법을 비교분석하였다. 연구 결과, 두 종류의 세균 모두 MRT-PCR법이 유의하게 높게 분석되었는데, 이는 죽은 세균의 DNA까지 분석되기 때문이었다. 따라서 MRT-PCR법에 의한 세균 총량방법은 구강 내 모든 균들을 대상으로 살아있는 균 뿐아니라 사멸 균까지 탐색할 수 있는 기술로서 임상적으로 매우 유용한 미생물 총량법이 될 것이다.