서 론
구강질환은 사람의 면역력, 당뇨병 등의 전신질환 유무, 흡연 및 음주 등과 관련된 전신적 요인 및 치은연하 치면세균막 내의 세균이 원인인 국소적 요인에 의해 발병 및 진행이 되는 것으로 알려져 있다[1,2]. 이들 대부분의 요인들은 구강 내 세균들이 주요한 원인인자로서 매우 밀접한 관계가 있다[3,4].
또한 구강질환 정도에 따라 병원성 세균의 종류나 그 총량의 차이를 보이며, 건강한 사람 간에서도 전신적 건강이나 면역성의 개별적 차이에 따라 통계적 유의성을 보이는 경우도 있다. 대부분의 치주질환균은 산소에 민감한 혐기성 세균들이기 때문에 치은연상 치면세균막에 비해 산소의 공급이 원활하지 않는 치은연하 치면세균막에 분포한다[5]. 1990년대 중반부터는 분자생물학의 비약적인 발전으로 이러한 원인균으로서 구강미생물 검출법은 25-100개의 세균만 있어도 검출이 가능한 중합효소연쇄반응(Polymerase chain reaction, PCR)이 사용되고 있다[6]. 중합효소연쇄반응은 세균 특이성이 있는 primer를 이용하여 적은 수의 세균이 있을지라도 쉽게 검출할 수 있는 유용한 방법으로서 이를 활용하여 구강 내 치태나 타액에서 직접 세균을 검출할 수 있게 되었다[7].
이러한 중합효소연쇄반응법도 세균의 존재여부를 확인하는 정성적인 방법에 국한되어 있어 정량적인 확인은 어려운 단점을 가지고 있지만, 최근에 real-time PCR이 확립되어 원인균의 존재여부를 확인하고 지수적 증식 단계를 조사함으로써 정량적 분석까지도 가능하다[8]. 특히 임상적인 미생물의 총량 분석방법은 실시간중합효소연쇄반응(Real-time polymerase chain reaction, RT-PCR)법으로 확립되었으며, 미국의 국립의학중앙도서관(National Library of MedicineNational Institutes of Health, NCBI)에 미생물의 DNA추출법과 Probe Method 등이 표준화여 등록되어 그 방법의 신뢰성이 확보되었다[9,10]. 이러한 Real-time PCR 검출법에도 SYBR Green quantitative polymerase chain reaction (SYBR Green qPCR)법과 프라이머 쌍 이외의 표적 핵산염기서열 특이 Probe을 첨가한 TaqMan법으로 나뉘기도 한다.
본 연구의 목적은 TaqMan법을 기초로 하여 동시적인 다균 검출법으로서 이 등[11]이 개발한 다중 실시간중합효소연쇄반응법(Multiplex real-time polymerase chain reaction, MRT-PCR) 시스템을 활용하여 타액 내 대표적인 치주질환과 치아우식증을 유발하는 구강질환균인 Prevotella intermedia (P. intermedia)와 Streptococcus mutans (S. mutans)의 총량과 모든 종류의 세균 총량을 대상으로 SYBR Green qPCR법(SYBR Green법) 및 단위부피당 형성된 집락 수법(Bacteria colony forming units, BC법)과 그 동등성 내지는 유효성을 비교 평가하였다.
연구방법
1. 타액 채취
본 임상 연구를 위해 C대학교 치과병원에서 IRB을 승인받아(No. CUDHIRB-1609-040) C대학교 치과병원에 내원한 18세에서 70세 사이의 전신건강이 양호한 70명을 선발하였고, 환자로부터 연구에 대한 설명과 동의를 받은 후 일련의 연구윤리의 규정에 따라 타액을 채취하였다. 이때, 환자의 나이, 성별, 과거력 및 현력, 흡연 유무 및 진단명 등을 제외한 어떠한 개인 식별 정보를 받지 않았다.
본 연구에 동의한 피검자에게 10 ml의 멸균된 증류수로 10초간 가글하고, 이를 50 ml conical tube에 뱉도록 하여 타액을 채취하였고, 이를 C대학에 소속한 한국구강미생물자원은행 실험실에 5분 안에 운반하여 일부는 세균을 배양하고, 나머지는 일반 PCR, SYBR Green qPCR 및 MRT-PCR을 시행하기 전까지 -80°C에 보관하였다.
2. Multiplex Real-time Polymerase Chain Reaction
TaqMan법을 기반으로 하는 MTR-PCR법은 새로이 PCR 과정 중 중합에서 사용되는 프라이머(primer) 및 프로브(이지페리오, YD Life Science, Gyeonggi, Korea)를 이용하여 시행하였다. 즉, 앞에서 채취한 타액 중 1 ml를 멸균된 에펜도르프 튜브(eppendorf tube, e-tube)에 옮긴 후 상온에서 소형 원심분리기를 이용하여 12,000 rpm에서 10분간 원심분리한 뒤, 침전된 세균(pellet)을 분리하였다. Pellet에 인산완충식염수(posphate buffered saline; PBS, pH 7.2) 1 ml 첨가 후 볼텍스(vortex)를 이용하여 혼합한 뒤 상온에서 원심분리(12,000 rpm, 10 min)한 후 상층액은 버리고 pellet을 얻었다. Pellet에 100 µl Direct PCR buffer (GeNet Bio, Daejeon, Korea)를 첨가하고 vortexing한 후 상온에서 10분간 반응시켰다(2분 간격으로 vortexing 함). 그 후 반응용액을 상온에서 소형 원심분리기를 이용하여 12,000 rpm에서 10분간 원심분리하여 상층액만을 취하여 타액 내 세균 검출을 위해 사용하였다. TaqMan법을 기반으로 한 MRT-PCR은 GeNet Bio사(Korea)의 HS prime qPCR premix (for TaqMan Probe)와 CFX96 Touch™ Real-Time PCR Detection System (Bio-Rad, Hercules, CA, USA)을 사용하여 실시하였다. 모든 세균을 검출하기 위한 표적 유전자는 16S rDNA 이었으며, 이 때 사용한 프로브는 “CY5”로 표기하고, P. intermedia와 S. mutans를 검출하는 데 사용한 프로브들은 각각 HEX와 FAM으로 표기하였다. MRT-PCR은 먼저 1 반응당 2xPremix 12.5 µl, 멸균수 7 µl, P.P mix 4 µl를 혼합하여 혼합액을 준비 한 후 혼합액 23.5 µl와 타액 내 세균 지놈 DNA 추출용액 1.5 µl를 혼합한 후 PCR을 진행하였다. 앞에 제시한 P.P mix는 forward primer와 reverse primer의 10 pmoles 그리고 Probe 5 pmoles을 각각 1 µl로 미리 혼합해 놓은 용액이다. PCR조건은 95°C에서 10분간 predenaturation 후, 95°C에서 15초 denaturation을 진행하고, 63°C에서 40초 결합(Annealing) 후 스캔(Scanning)하는 과정을 44번 반복하였다. 이때 사용한 프라이머는 <Table 1>과 같다.
|
Table 1. Nucleotide sequence and reaction conditions of real-time quantitative PCR primer using MRT-PCR method 
|
*Prevotella intermedia, **Streptococcus mutans
3. SYBR Green법을 이용한 qPCR
멸균 증류수(10 ml)로 가글한 타액 샘플은 얼림(-80°C)과 녹임(25°C)을 세 차례 반복하고 이를 10분간 100°C에서 끓여서 qPCR을 할 수 있도록 준비하였다. qPCR은 TOPreal qPCR 2 × PreMIX (SYBR Green) kit (Enzynommics, Daejeon, Korea) 및 ExicyclerTM 96 real-time quantitative thermal block를 사용하여 실시하였다. qPCR에 사용될 프라이머는 Park 등[12,13]이 개발한 것을 사용하였으며<Table 2>, 이는 Bioneer사(Daejeon, Korea)에 의뢰하여 제작하였다. qPCR 반응은 PCR PreMix에 forward 및 reverse 프라이머 각각 10 pmoles 1 µl와 세균 유전체 DNA 또는 타액 내 세균 DNA 2 µl를 PCR tube에 넣고, PCR PreMix kit에 동봉된 DEPC water를 넣어 최종 반응물이 20 µl가 되도록 한 후 충분히 voltexing하였다. 정량 실시간 PCR 조건은 95°C에서 10분간 predenaturation 하고 95°C에서 10초간 denaturation 한 후, P. intermedia와 total 세균의 검출을 위해서 60°C에서 20초간 결합(Annealing), 72°C에서 20초간 extension하고, S. mutans세균의 검출을 위해서는 70°C에서 30초간 annealing과 extension하고, scanning하는 과정을 40회 반복하였으며, melting curve 분석은 60~94°C에서 1°C씩 1초간 시행하였다. 이상의 qPCR반응은 3회 반복 실시하여 평균하였다. 이때 Standard curve와 최소검출 한도를 측정하기 위해 각 균주의 유전체 DNA를 20 ng부터 2 fg까지 10단계씩 희석하여 실험하였다.
|
Table 2. Nucleotide sequence and reaction conditions of real-time quantitative PCR primer using SYBR Green method 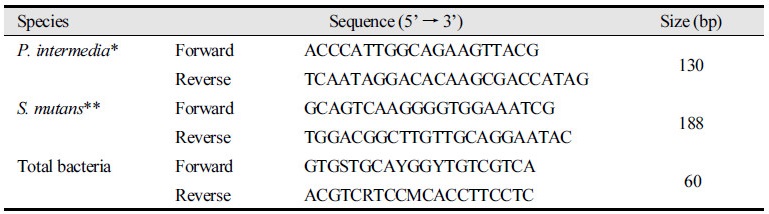
|
*Prevotella intermedia, **Streptococcus mutan
4. 세균 배양
타액의 세균을 배양하기 위해 Tryptic Soy broth (Difco Laboratories, Detroit, MI, USA)에 0.5% yeast extract, 0.05% cysteine HCl-H2 O,0.5 mg/mlhemin 및 2 µg/ml vitamin K1가 포함된 배지(TSB-YCHVk1 배지)를 사용하였다. 특히 S. mutans를 선택적으로 배양하기 위해서 mitis salivarius agar (Difco Laboratories)에 0.0001% potassium tellurite, 0.2 units (2.8 µg/ml) of bacitracin (Sigma Chemical Co., St. Louis, MO, USA) 및 20% (w/v) sucrose (CJ Co., Seoul, Korea)가 첨가된 배지를 이용하였다. S. mutans는 37°C incubator에 배양하고, 나머지 균주들은 37°C anaerobic chamber (Bactron I, Sheldon Manufacturing Inc., Cornelius, OR, USA)의 혐기성 조건(10% H2, 5% CO2, 85% N2)에서 배양하였다.
모든 세균 및 P. interemdia를 배양하기 위해 멸균 증류수 (10 ml)로 가글한 타액 용액 중 10µl를 인산완충식염수(posphate buffered saline; PBS, pH 7.2)로 10배, 100배 희석하여 TSB-YCHVk1 한천 배지에 도말한 후 혐기성 배양기에서 5-7일 동안 배양하였다. 또한, S. mutans를 배양하기 위해, 멸균 증류수 (10 ml)로 가글한 타액 용액 10 µl 및 100 µl를 S. mutans 선택배지(한천 배지)에 도말하여 실온에서 1일 배양하고, 이를 다시 37°C incubator에서 2-3일을 배양하였다.
5. P. intermedia 와 S. mutans 의 검출을 위한 군락 PCR
앞에서 배양한 배지에서 P. intermedia와 S. mutans의 검출을 위해 군락의 색깔 및 모양을 관찰하고, 각각의 세균 종이라 생각되는 군락들을 채취하여 50 µl 인산완충식염수에 혼탁하였다. 종-특이 PCR 프라이머를 이용하여 일반 PCR 또는 SYBR Green qPCR을 한 후, PCR 증폭물을 1.5% 아가로스 젤에 10 µl를 전기영동 하여 그 증폭물의 존재여부와 크기로 해당 종에 속하는지를 판정하였다. 이때 사용한 프라이머는 <Table 3>과 같다.
|
Table 3. Nucleotide sequence and reaction conditions of PCR primers for cluster PCR 
|
*Prevotella intermedia, **Streptococcus mutans
6. 세균 검출 방법에 따른 검출된 총 세균 수 비교
세균 검출법 간에 어느 방법이 타액 내 총 세균 수를 더 잘 검출하는지를 알아보기 위해 국 등[7]이 개발한 “세균 검출 방법 간 총 세균 검출 지수”를 다음과 같은 식(formula)을 이용하여 구하였다.
만약 “B 방법 대비 A 방법의 총 세균 검출 지수”가 “5”인 값을 갖는다면, 이는 A 방법에 의해 타액 내 세균 총 검출 수가 B 방법에 의한 것보다 5배가 더 많았다는 것을 의미하며, 이는 “세균 검출 간의 타액 내 모든 세균 수를 검출할 수 있는 정도 차이의 척도로 사용”될 수 있다. 또한, “세균 검출 방법 간 총 세균 검출 지수”는 두 방법 간의 타액 내 총 세균 검출 일치도를 나타내기도 한다. 즉 “세균 검출 방법 간 총 세균 검출 지수”가 “1”에 가까울수록 두 비교 방법들에 의한 세균 검출 수가 서로 일치한다는 것을 의미한다.
연구결과
1. 타액의 채취
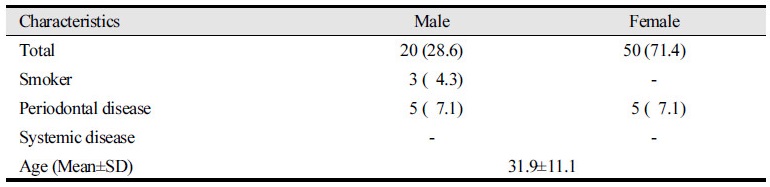
연구대상은 C치과병원에 내원한 18세에서 70세 사이의 전신건강이 양호한 70명이었다. 이는 본 의료기관내 연구윤리위원회의 규정에 제시된 규칙 내에서 피검자 선발과 동시에 타액 검체를 수집하였다. 채취한 피검자들의 일반적인 특성은 남성 28.6%, 여성 71.4%이었고, 흡연자는 남성에서만 4.3%로 나타났고, 치주질환자는 남녀 각각 7.1%로 조사되었다<Table 4>.
2. MRT법, SYBR Green qPCR법 및 선택배양법을 이용한 세균검출 평균량 및 세균 수 비교
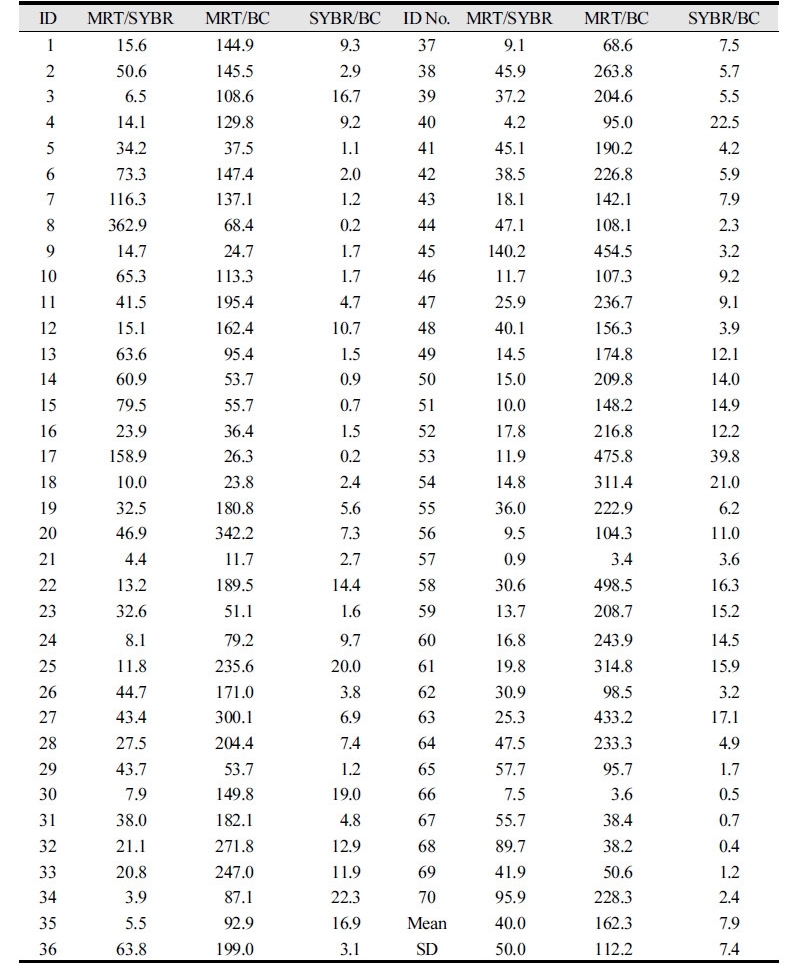
피검자 MRT법, SYBR Green 및 선택배양법에 의한 타액 내 전체 세균과 S. mutans 및 P. intermedia의 평균 총량을 비교한 결과는 <Table 5>와 같다. MRT-PCR법에서는 S. mutans가 50341.309×103 cells, P. intermedia가 450571.381×103 cells로 측정되었고, SYBR Green qPCR법으로는 S. mutans가 14344.871×103 cells, P. intermedia가 119.639×103 cells로 측정되었으며, BC법에서는 1.791×103 cells, P. intermedia가 735.857×103 cells 로 측정되었다. MRT-PCR법과 SYBR Green qPCR법의 세균 검출 비율을 분석한 결과 MRT-PCR법에 의해 약 40배(0.9배에서 362.9배)정도 더 많은 세균이 검출되었음을 알 수 있었다. 그러나 타액 내 P. intermedia와 S. mutans의 검출 빈도는 세 방법 간의 차이가 6.23배 내지 1,164.35배 차이가 나고, 실제 타액 내 P. intermedia와 S. mutans의 존재 비율을 알 수 없기 때문에 세 방법들 중 어느 방법이 가장 뛰어나는 지에 대한 비교는 큰 의미가 없다.
|
Table 5. Total amount of bacteria in saliva by MRT method, SYBR Green and BC method (Unit: ×103 cells, N=70) 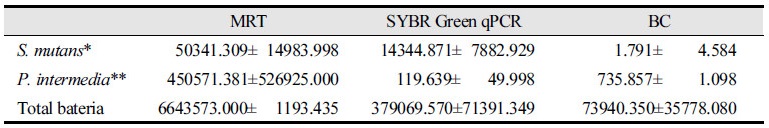
|
*Streptococcus mutans, **Prevotella intermedia
또한 MRT-PCR법과 세균배양법간의 상대적 검출량 비교 시, MRT-PCR법은 세균배양법에 비해 약 162.3배(3.4-498.5배) 정도 더 많은 세균이 검출되었다<Table 6>. SYBR Green qPCR법과 세균배양법을 비교하면, SYBR Green qPCR법이 세균 배양법보다 약 7.9배(0.2배 내지 39.8배) 정도 더 많은 세균을 검출할 수 있었다. 이러한 결과는 MRT-PCR법 > SYBR Green qPCR법 > 세균배양법 순으로 타액 내 세균을 더 많이 검출할 수 있다는 것과, MRT-PCR법과 SYBR Green qPCR법 간의 타액 내 총 세균 검출 결과가 가장 일치함을 의미한다.
3. 세균 검출 방법에 따른 P. intermedia 및 S. mutans 검출 유무 비교
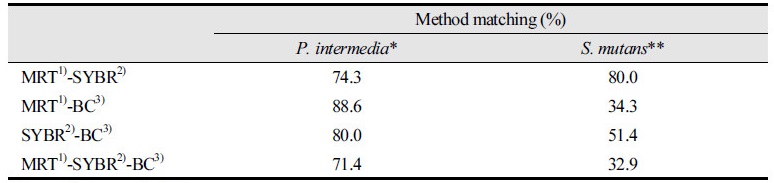
세 가지 방법에 따른 P. intermedia와 S. mutans의 검출 유무는 <Table 7>에 제시하였다. MRT- PCR법에 의해서는 모든 타액 샘플에서 P. intermedia와 S. mutans가 검출되었지만, SYBR Green qPCR법과 세균 배양법에 의해서는 P. intermedia 또는 S. mutans가 검출되지 않은 경우도 있었다. 세 가지 방법 모두에서 P. intermedia 및 S. mutans가 검출된 경우는 각각 71.4%와 32.9%였다<Table 7>.
|
Table 7. The presence of P. intermedia and S. mutans detection among the three meth 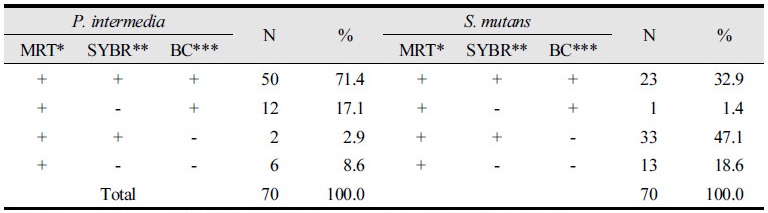
|
*Multiplex real-time quantitative PCR; **SYBR Green quantitative PCR; ***Bacterial culture
P. intermedia 검출에 있어서 세 방법들 간의 일치도는 71.4 ~ 88.6%로 비슷한 편였으나, S. mutans의 경우 MRT-PCR법과 SYBR Green qPCR법 간의 일치도는 80%이었지만, MRT-PCR법과 세균배양법 간은 34.3%, SYBR Green qPCR법과 세균배양법 간은 51.4%였다<Table 8>.
총괄 및 고안
전통적으로 세균배양법은 선택적 배지를 활용하여 특정세균만을 배양하는 방법으로서 미생물 분석 표준 정량법으로 알려졌다[12]. 그러나 세균 배양법은 유사종과 속의 외형만으로는 잘못된 결과를 얻을 수 있었고, 또한 현재까지 구강미생물 700여 종 가운데 20여 개 정도만 선택배지가 규명되었을 뿐이다[13,14]. Real-time PCR법에 의한 정량분석은 구강 내 사멸된 균에서도 DNA 추출 정량인 만큼 일반적으로 살아있는 미생물을 대상으로 한 선택배지 군락형성으로 인한 균정량 법보다 많은 균량이 검출되는 것이 일반적이다[15]. 구강 내 사멸 균 정량 또한 중요한 요인으로 치주질환원인균 특성상 혐기성세균으로서 자신의 세포외막에 강력한 독성물질인 지질다당체(lipopolysaccharide, LPS) 등으로 둘러 쌓여 있으며[16], 이들 세포의 부속물질들도 치주조직에 박히거나 염증물질을 방출하여 치주질환을 야기하는 데 있어서 살아있는 균만큼의 독성을 유지하기 때문이다. 하지만 여전히 세균배양법이 유용하게 사용되고 또한 임상결과로서 과학성이 유효하게 인정되고 있어서 이번 연구는 MRT-PCR법의 유효성을 확인하고자 함이지만, 이의 정확성이나 유효성을 평가하기 위해서는 세균배양법 그리고 SYBR green qPCR법 등과의 비교가 필요하다.
따라서 이미 선택배지로 균 배양법이 정립된 구강 미생물 가운데 대표적인 S. mutans와 P. intermedia를 정량화하기 위해 대상균으로써 선택하였다. 정량적 분석을 위해 선택배지법, 전통적인 SYBR green qPCR법, 그리고 이들과 비교하기 위해 새로이 개발된 프라이머 및 프로브((주)와이디생명과학, 경기, 한국)를 이용한 TaqMan법을 기반으로 한 MRT-PCR법과의 일치성 혹은 동등성을 평가하고자 했다. 그 결과 본 연구에서 비교한 세 가지 세균 검출법들 중에서 MRT-PCR법에 의해 타액 내 총 세균, P. intermedia 및 S. mutans 세균 수가 SYBR Green qPCR법 및 세균 배양법보다 더 높게 측정되었다.
타액 내 P. intermedia 및 S. mutans 검출 유무만을 비교할 때, 방법 간 일치도는 P. intermedia의 경우 MRT-PCR법과 세균배양법이 가장 높은 일치도(88.6%)를 보였고, S. mutans의 경우 MRT- PCR법과 SYBR Green qPCR법이 가장 높은 일치도(80.0%)를 보였다. 타액 내 P. intermedia와 S. mutans의 검출 빈도는 세 방법 간의 차이가 6.23-1,164.35배 차이가 나고, 실제 타액 내 P. intermedia와 S. mutans의 존재 비율을 알 수 없기 때문에 세 방법들 중 어느 방법이 가장 뛰어나는지에 대한 비교는 큰 의미가 없다고 판단된다.
그러므로 타액 내 총 세균, P. intermedia 및 S. mutans 수를 가장 많게 측정하고, 방법 간 P. intermedia 및 S. mutans 검출 유무 일치도를 고려할 때, MRT-PCR법이 타액 내 구강 세균 종 검출에 비교적 가장 우수하다고 생각된다. 이와 유사한 결과로 치태 내 미생물을 배양과 qPCR에 대한 방법을 이용하여 특정 세균 검출을 비교한 연구를 통해 배양법과 qPCR이 유의한 차이를 나타낸다고 하였으며[17], 근관 및 치주 미생물학에 대한 배양과 PCR을 비교하여 특정세균의 검출이 배양보다 PCR법이 더 민감하고 높은 신뢰성이 있다는 보고가 되어있다[18,19].
또한 Yano 등[20]에 따르면 치아우식 균인 S. mutans를 real-time PCR법과 배양법을 통해 정량 분석한 결과, PCR법이 더 높은 정량을 가지는 것을 확인하였고, 치주질환 균 P. intermedia는 real-time PCR법과 배양법을 비교한 결과 정량 값이 일치하지 않았다는 연구결과도 있다[21]. Verner 등[22]의 연구결과에 따르면 Real-time PCR과 배양법을 비교한 연구에서도 배양에서는 산소에 민감한 T. denticola를 검출하지 못했지만 Real-time PCR에서는 검출이 되었으며 그만큼 형광 첨자를 이용하여 세균검출 수준이 상당히 높다. 이번 결과에서도 앞선 연구와 동일한 결과를 얻었으며, 차이가 나는 부분에 대한 이유로 배양에서는 산소에 민감한 혐기성세균들일 경우 자라지 못하는 경우, qPCR에서는 죽은 세균까지 추적되기 때문에 이와 같은 이유로 판단되며 죽은 세균의 세포막에서 세포외막의 독성, 세포날개 등이 치주조직에 염증을 지속적으로 야기시키기 때문에 죽은 세균의 정량 또한 중요하다[23].
이상의 연구는 치주질환 원인균과 충치 원인균을 MRT-PCR을 이용하여 정량 분석하는데 그 유효성을 세균배양법과 qPCR법인 SYBR Green법 등과 비교한 결과, 선행연구에서도 보고되었듯이 MRT-PCR방법의 이점으로써 사멸된 균까지 추가로 추적되어서 사멸 균의 위험성까지 경고할 수 있었으며 또한 MRT-PCR의 경우 한 번의 반응으로 한꺼번에 5종 세균을 검출할 수 있어 SYBR Green qPCR법에 비해 최대 5배의 시간을 단축시킬 수 있는 장점이 있다. 더욱이 최근까지도 인정되는 전통적인 세균배양법의 경우, 혐기성 세균 배양기간이 최대 7-10일까지 소요되는 점을 볼 때, MRT-PCR법이 매우 유용하다는 것을 확인할 수 있었다. 또한 특정 세균 종을 검출하기 하기 위해 선택배지를 사용하더라도 100% 특정 세균 종을 선택적으로 배양할 수 있는 배지는 없기 때문에 배양된 군락의 세균으로부터 16S rDNA 유전자를 클로닝하여 시퀀싱하거나 본 시험에서처럼 종-특이 PCR 프라이머를 이용하여 군락 PCR을 시행해야 함으로 많은 시간과 비용이 필요하다는 걸 한 번 더 확인하였다.
비록 본 연구는 한 기관에서 진행된 단편적인 연구였으며, 치주질환자와 정상인을 구분하여 균의 총량이나 균 종류의 분포 등을 파악할 수 있는 단계의 디자인이었다면 좀 더 활용단계가 높은 유용한 결과를 도출할수 있었을 것이라는 아쉬운 점이 남는다. 향후 연구 디자인에서 치은염과 치주질환자의 병기별로 균의 총량 및 균종의 분포 등을 확인하고자 하는 임상적 연구 디자인을 시도해 보고자 한다. 이러한 후속연구의 필요성에도 불구하고 본 연구의 목적으로서 TaqMan법을 기초로 하여 동시적인 다균 검출법으로서 이 등[11]이 개발한 MRT법 시스템을 활용하여 타액 내 대표적인 구강질환균 P. intermedia와 S. mutans의 총량과 모든 종류의 세균 총량분석법으로서 매우 유효하고 정확한 연구였다. MRT-PCR법을 통해 향후 구강미생물의 정량적 정성적 분석에서는 고전적이고 배양할 수 있는 세균이 수종에 불과한 BC법 및 SYBR Green qPCR보다 시간과 비용을 단축할 수 있는 MRT-PCR법의 이점을 활용하여 의료분야에서는 10여 년 전부터 진단방법 중의 하나로 인정되어 왔으며, 앞으로 MRT-PCR법은 치주질환과 치아우식증 등 구강질환의 주요원인 균을 확인하는 데 있어 매우 유용한 정량 및 정성 검사에 상당한 도움이 될 것이라 판단된다.
결 론
본 연구는 새로이 개발된 TaqMan법 응용인 MTR-PCR법을 기존의 선택 배지를 구강 미생물 배양법, SYBR Green qPCR 등과 정량적 비교분석연구를 통하여 정확성 일치성 등 유효성을 확인하고자 했다. SYBR Green 방법은 경구 용 타액 내 구강 미생물의 총량을 분석하는 데 널리 사용되지만, 본 연구에서는 새로 개발 된 프라이머와 프로브를 이용하여 TaqMan 방법에 기반한 MTR-PCR법의 정확성을 목표로 연구설계되었다. 연구결과, MRT-PCR법과 SYBR Green qPCR법의 세균 검출 비율을 분석한 결과, MRT-PCR법에 의해 약 40배(0.9-362.9배) 정도 더 많은 세균이 검출되었음을 알 수 있었으며, MRT-PCR법 > SYBR Green qPCR법 > 세균배양법 순으로 타액 내 세균을 더 많이 검출 되었었다. 또한, MRT-PCR법과 SYBR Green qPCR법 간의 타액 내 총 세균 검출 결과가 가장 일치도가 높았으며, P. intermedia 검출에 있어서 세 방법들 간의 일치도는 71.4~88.6%로 비슷한 편였으나, S. mutans의 경우 MRT-PCR법과 SYBR Green qPCR법 간의 일치도는 80%였다. 타액 내 P. intermedia 및 S. mutans 검출 유무만을 비교할 때, 방법 간 일치도는 P. intermedia의 경우 MRT- PCR법과 세균배양법이 가장 높은 일치도(88.6%)를 보였고, S. mutans의 경우 MRT-PCR법과 SYBR Green qPCR법이 가장 높은 일치도(80.0%)를 보였다. 연구결과 아래와 같은 결론을 얻었다.
1.MRT-PCR법에 의해 타액 내 총 세균, P. intermedia 및 S. mutans 세균 수가 SYBR Green qPCR법 및 세균 배양법보다 더 높게 측정되었다.
2.MRT-PCR법 > SYBR Green qPCR법 > 세균배양법 순으로 타액 내 세균을 더 많이 검출되었으며, 이는 MRT-PCR법이 가장 정밀하였다.
3.방법 간 일치도는 P. intermedia의 경우 MRT-PCR법과 세균배양법이 가장 높은 일치도(88.6%)를 보였고, S. mutans의 경우 MRT-PCR법과 SYBR Green qPCR법이 가장 높은 일치도(80.0%)를 보였다.
4.MRT-PCR법이 다른 정량분석법보다 시간적 경제적 유효성이 장점으로 나타났다.
결론적으로 본 연구를 통하여 사람 타액 내 존재하는 다균 종을 정확하고 신속한 검출하는 방법으로써 MRT-PCR법이 향후에 널리 사용될 것으로 예측된다.